 |
1.
Découverte
des fonctions principales
Fichiers . pdb rasmol utilisables sur internet : iggtotal.pdb (691Ko) igg-lys.pdb (287Ko) |
 |
1.
Découverte
des fonctions principales
Fichiers . pdb rasmol utilisables sur internet : iggtotal.pdb (691Ko) igg-lys.pdb (287Ko) |
| TD informatique
, 2 heures, en BTS AB 1, Lycée Jolimont Toulouse.
Ouvrir le fichier
: RASMENUF.EXE :
SOMMAIRE du TD : 1. Travail sur la molécule entière d'IgG 1. 2. Travail sur le site
anticorps d'un fragment Fab associé
à l'antigène correspondant (lysozyme) .
|
1.
Travail à réaliser sur le fichier iggtotal.pdb :
Afficher
: sphères : rayon de Van der Waals.
Observer
la molécule, la faire tourner selon différents angles.
Pour se
repérer dans l'espace, utiliser l'option : système d'axes.
|
|
|
|
|
|
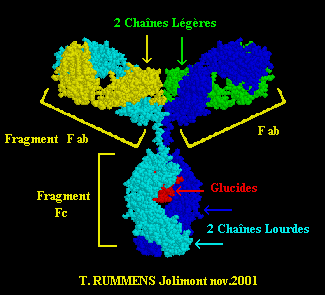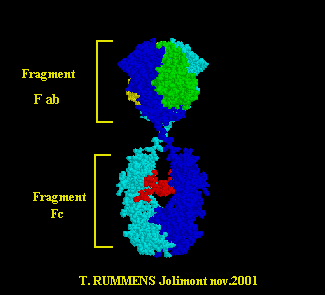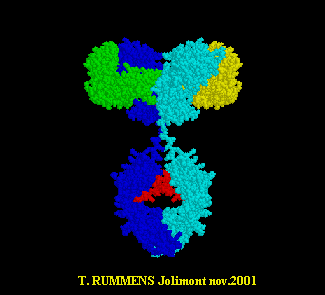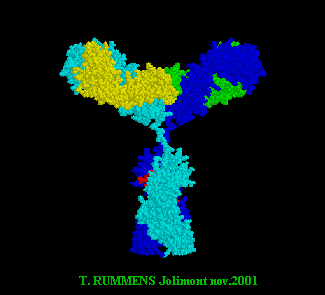
Bleu clair et foncé : chaînes lourdes H. Rouge : la chaîne glucidique. |
de 90 ° / axe vertical. |
Ecrire : les premières observations ( constatations / structure de l' IgG 1 ) .
Pour différencier les différentes
chaînes : colorer par chaîne et segment.
Faire varier l'affichage avec :
bâtonnets, sphères, chaîne carbonée, rubans.
|
|
|
|
|
|
|
|
Ecrire les
observations nouvelles / structure de l' IgG 1.
Réaliser
un dessin simplifié illustrant vos observations en le légendant.
NB : le
dessin simplifié doit suggérer au lecteur la structure de
la molécule dans l'espace.
Afin d'affiner la découverte,
l'étude et la représentation de l'architecture de la molécule,
utiliser les options suivantes avec
rasmol :
Afficher
: squelette carboné ou rubans, et colorer par chaîne.
Afficher
les ponts disulfure, les colorer en rouge,
et
options : ponts disulfure : squelette carboné afin de mieux les
visualiser.
|
|
(bleue claire, et bleue foncée) à la base de séparation des 2 bras de la forme en lettre Y. Un pont est
situé entre chaque chaîne H et la fin de chaque
On devine d'autres
ponts disulfure qui sont situés à l'intérieur
Il convient
de vérifier la position des cystéines dans la séquence
|
Préciser
alors sur un schéma très simplifié de la structure
IV de la molécule d' IgG 1 les éléments suivants :
la nature des chaînes protéiques,
le nombre de ponts disulfure et leur position exacte sur chaque chaîne,
leur situation : pont intra-chaîne ou pont inter-chaîne,
les extrémités NH2 et COOH terminales de chaque chaîne.
Eventuellement la nature des acides aminés de la chaîne lourde
H ( Heavy ) qui lui permettent
de se courber avant de s'associer avec la chaîne légère
L ( Light ).
Afficher les liaisons hydrogènes.
Quelle constatation
et quelle conclusion peut-on alors établir quant à la structure
IV de la molécule ?
Comment
la cohésion des domaines globulaire est-elle assurée ?
|
|
|
| Les domaines
en bêta feuillets sont très nombreux
et sont regroupés en 4 "globules" par chaîne lourde et en 2 "globules" par chaîne légère. |
Les liaisons
H en violet, sont très nombreuses et
permettent de relier entre-eux les bêta feuillets afin de renforcer la cohésion de l'ensemble des "globules". |
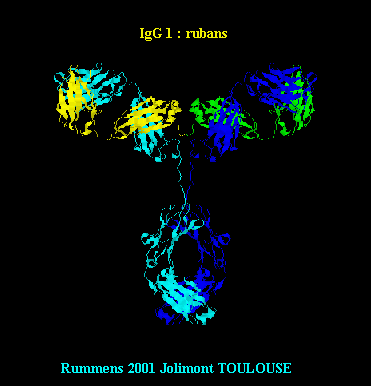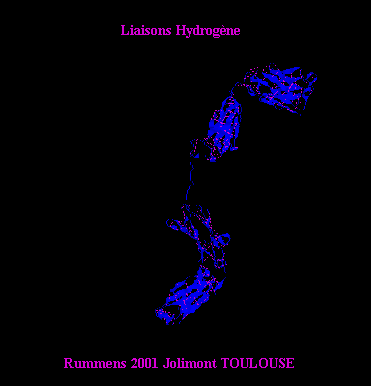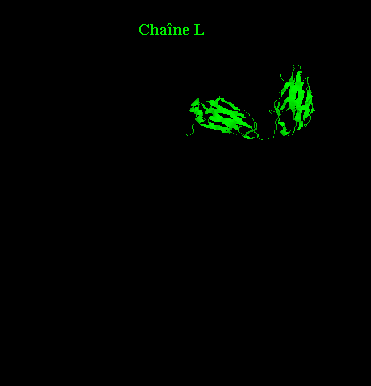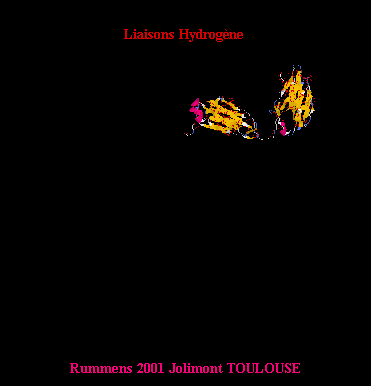 |
1 : Vue globale des 4 chaînes, en rubans. 2 : Chaîne lourde H, en ruban. 3 : Chaîne H et liaisons Hydrogène en violet. 4 : Chaîne H avec feuillets, hélices et boucles. 5 : Chaîne L, en rubans. 6 : Chaîne L avec feuillets, hélices et boucles. 7: Chaîne
L avec liaisons Hydrogène en rouge.
|
Séquence de la chaîne
légère L : on peut renuméroter les acides aminés
en sachant que n° 1 = Glu 501.
Ainsi une chaîne L, est composée
de 216 acides aminés.
2.
Travail à réaliser sur le fichier igglys.pdb : fragment Fab
et lysozyme d'œuf.
Ouvrir le fichier igglys.pdb.
Il correspond à un fragment
Fab de l'anticorps IgG 1 avec le site anticorps anti lysozyme d'œuf.
L'Ac et l'Ag sont ici représentés.
Afficher : rubans ou chaîne carbonée. Colorer les différentes
chaînes.
Reconnaître le site anticorps
du fragment Fab.
Reconnaître l'antigène
( la molécule de lysozyme d'œuf ) .
A l'aide
du clic de la souris, établir une liste de 6 acides aminés
pour l'antigène d'une part,
et pour
le site anticorps d'autre part.
Formuler
par écrit, la nature des liaisons chimiques qui pourraient s'établir
entre les AA de l'antigène
et les
AA du site anticorps ainsi répertoriés, liaisons qui pourraient
expliquer la façon
dont le
site anticorps pourrait reconnaître le déterminant antigénique
porté par le lysozyme.
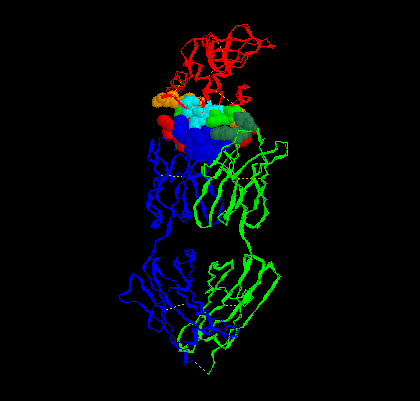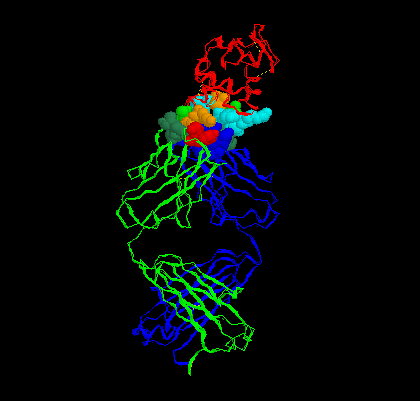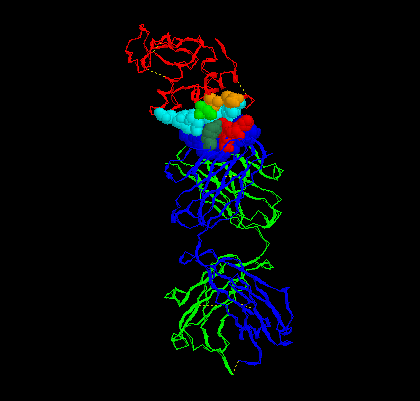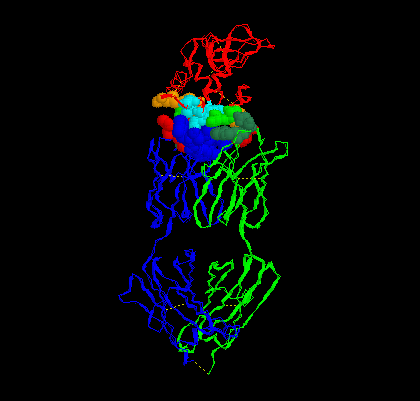 |
Dans le menu principal,
écrire la ligne de commande :
script sites.spt Observer ce qui est apparu : une représentation en sphères des acides aminés qui entrent en contact lors de la reconnaissance anticorps / antigène. En faisant tourner la molécule, établir la liste des AA de l'antigène et celle du site anticorps, qui entrent en contact. On peut modifier l'affiche
du "script" afin de reconnaître la
La
comparer avec la liste déjà établie,
Représenter
de façon simplifiée l'ensemble Antigène Anticorps
Fab après avoir choisi une orientation appropriée de
|
4. Bibliographie.
| Rasmol , logiciel créé
par :
Glaxo Wellcome Research
|
Structures et fonctions des molécules biologiques Utilisations pédagogiques
|
institut national de recherche pédagogique 29 rue d'Ulm,
|